알림
이 포스팅은 2019년 Harvard STAT115강의를 요약한 것임을 알려드립니다.
다룰 토픽
- SNP, LD, haplotypes, tagging SNPs
- GWAS: Family-based, Case-control
- Population stratification
- eQTL: differential expression와 관련된 SNP를 identify하기
- cell-type specific expression : 질병 관련 tissue를 identify하기
Polymorphism (다형성)
네이버 지식백과에 따르면 ploymorphism은 같은 종의 생물이지만, 모습이나 고유한 특징이 다양한 성질을 말한다. 즉 염기서열의 common variation을 가지는 site(위치) 혹은 gene(유전자)을 의미한다. 이러한 작은 variation이 표현형에서는 큰 변화를 가져오기도 한다 (예: 인종별 차이). 몇 가지 용어를 정의해보자.
- locus : genome (유전체, DNA로 구성된 유전 정보의 총체) 내의 위치 (location) 를 의미한다. gene 전체가 될 수도 있고 (예를 들면, 1번 염색체), 하나의 position을 의미할 수도 있다.
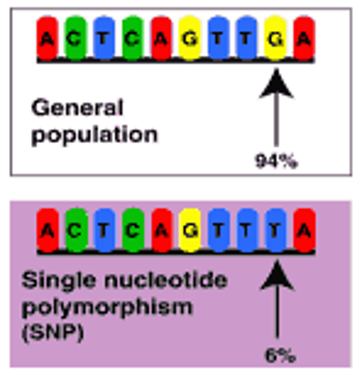
- alleles : 유전자의 변형 (variation)을 의미한다. 예를 들면, 일반적인 population에서의 염기서열이 ACTC-AGTT-GA이라면, SNP (Single nucleotide Polymorphism)에서의 염기서열은 ACTC-AGTT-TA로 G대신 T로 variation이 발생하였다. 이때 이에 해당하는 locus에서 G/T alleles가 있다고 말한다.
- MAF (Minor Allele Frequency) : MAF 1% 이상일 때 ploymorphic하다고 한다. 그 이하로 variation이 발생하는 것은 ploymorphic하다고 하지 않는다. 아래 왼쪽의 그림에서 T의 variation은 6% 이므로 ploymorphic하다고 할 수 있다.
- SNP (Single nucleotide Polymorphism) : DNA 복제 과정에서 생기는 유전자 염기서열의 변이로, 생식세포 (정자, 난자)에서 mutation이 발생하여 다음 세대로 유전된다. 사람들 간의 표현형의 차이는 ~90% 정도가 이 SNPs (유전자 변이)에 기인한 것이다.

Linkage Disequilibrium (LD): SNP의 특징
Hardy-Weinberg equilibrium (HWE)
- genotype AA, aa, Aa를 가지는 population에서 라 하고, 라고 하자.
- 그러면, , , , where 이다.
- A와 B가 독립이라는 가정 하에, loci가 2개인 경우 A/a와 B/b를 가지는 Hardy-Weinberg equilibrium은 다음과 같다. (가정 : A와 B가 독립)
- P(A) = 0.6, P(a) = 0.4, P(B) = 0.6, P(b) = 0.4일 때, P(AB) = 0.6 * 0.6, P(Ab) = 0.6 * 0.4, P(aB) = 0.4 * 0.6, P(ab) = 0.4 * 0.4.
- 하지만, 이러한 HWE를 따르지 않는 경우도 있다. 이는 allele A와 allele B의 발생이 독립적이지 않기 떄문이다. (LD, Linkage Disequilibrium)
LD (Linkage Disequilibrium)
- allele들이 우연보다 더 자주 동시에 발생하는 현상. 이는 DNA 상에서 두 allele가 물리적으로 가깝게 위차해있기 때문에 발생한다.
haplotype block
- 강한 LD를 가지는 서로 연관된 (linked) SNP들의 클러스터를 의미한다 (아래 그림의 빨간 삼각형). 따라서, 한 haplotype block내의 allele를 predict할 수 있으면, LD에 의해 다른 allele들도 예측할 수 있다.
- 해플로타입 block의 경계는 재조합 (recombination)이 잘 발생하는 지역이다.

- 해플로타입을 이용한 association studies는 individual SNP들을 이용한 경우보다 더 정확하다.
SNP profiling
[C/T][A/G]T X C[A/C][T/A]와 같은 염기서열이 있다고 하자. 그러면 가능한 해플로타입 조합의 수는 개, 즉 16개가 있다. 하지만 실제로는 많은 SNP들이 연관 (linked) 되어있기 때문에 16개보다 훨씬 더 적은 3~4개의 조합이 존재하게 된다. 그리고 이러한 3~4개의 조합이 90%의 유전적 variation의 이유가 된다.
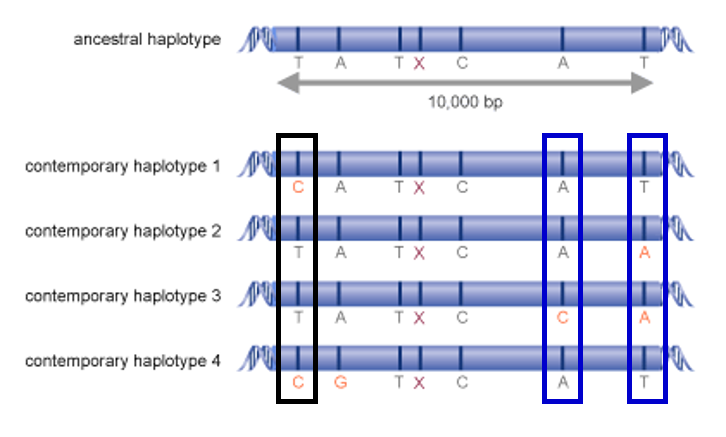
Tagging SNPs (non-redundant, unique)
- 이러한 연관성 덕분에, 모든 가능한 조합의 염기서열을 알 필요 없이 일부 non-redundant (비중복적인)한 염기서열들만 알게되면 다른 나머지 가능한 염기서열을 자동으로 알게 된다. 이러한 비중복적인 염기서열들을 tagging SNPs라고 한다.
- Tagging SNPs들은 해플로타입내의 변이를 대부분 설명한다.
- Tagging SNPs들은 Reference SNP ID number (예: rs12345678) 을 가지고 있다.
- 위의 그림에서 ancestral haplotype은 16개의 가능한 조합을 가진다. 그 중에 연관성에 의해 실제 존재하는 조합이 4개라고 할 때 (contemporary haplotype 1 ~ 4), 그 중 3개만 알고 있으면 나머지 한개는 예측할 수 있다. 이때 알고있는 3개의 haplotype을 tagging SNPs라고 한다.
과거의 전체 genome의 SNP array는 이러한 과정으로 완성되었다. (전체를 분석한 것이 아니라, 일부만 분석한 후 연관성에 의해 나머지 부분을 예측하는 방식이 사용되었다.) 현재에는 genome sequencing의 가격이 내려가고 기술이 발전함에 따라 WES (Whole Exome Sequencing)이나 WGS (Whole Genome Sequenciing)등 전체 sequence를 분석한다.
Genome-Wide Association Studies (GWAS)
1) Association Study
Association study는 Genetic markers와 phenotype (표현형) 사이의 연관성을 연구한다. 예를 들면, 낭성 섬유증 (Cystic Fibrosis) 환자의 70%는 3개의 염기쌍이 deletion 되어있다. 이는 CFTR gene의 508번 position에서 페닐 알라닌 아미노산을 만들지 못하는 것이 원인이라고 할 수 있다. Association study에서는 특히 질병 유전자를 찾거나, SNP / haplotype marker을 찾거나, 특정 질병에 대한 개인의 취약성을 파악하는데 목적을 두고 있다.
2) GWAS를 수행하기 전 먼저 Quality Control을 해야 한다.
- Genotyping quality를 확인한다.
- 잘못된 성별이 있는지 확인한다.
- individual간 비정상적인 유사성 (sample swap, 혹은 중복샘플, 쌍둥이)이 있는지 확인한다.
- Family-based association study를 수행할 경우 Mendelian 유전의 법칙에 Trio (부,모,자녀)가 잘 부합하는지 확인해야 한다.
3) GWAS에는 두 가지 방식이 있다.
- Family-based association studies (과거에 많이 연구됨)
- Population-based case-control association studies (최근 많이 연구됨, 보편적)
Family-based association studies
아래 왼쪽그림은 멘델의 유전법칙에 따른 부, 모, 자녀의 genotype을 보여준다. 오른쪽 그림은 염기 A/a의 transmit을 표로 나타낸 것인데 a와 d는 항상 0이다. 왜냐하면 transmitted 되고, 동시에 non-transmitted되는 상황은 발생하지 않기 때문이다. 이 표를통해서 a보다 A가 훨씬 많이 transmit 되거나, 그 반대의 상황이 발생하는지 확인해볼 수 있다.
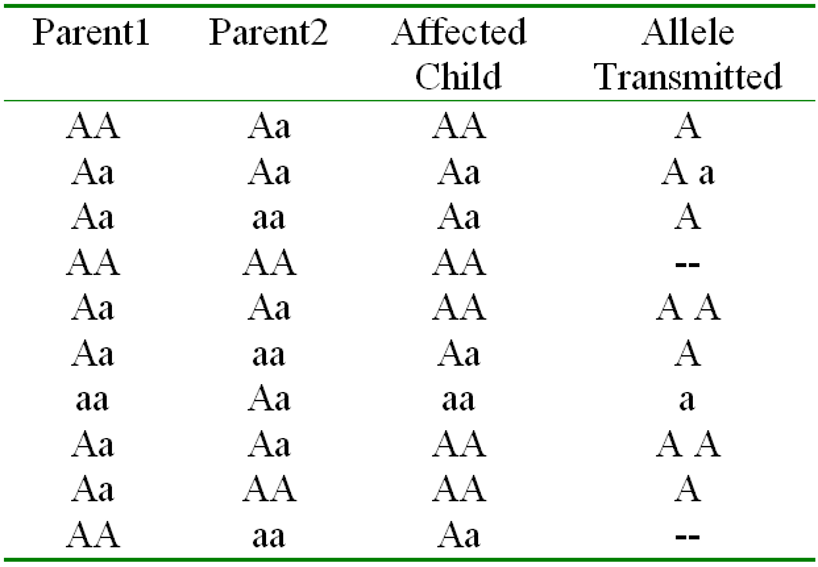
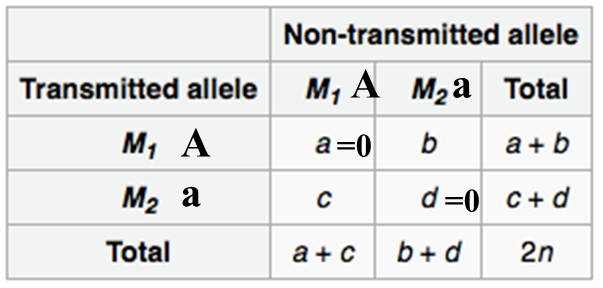
TDT (Transmission Disequilibrium Test)
- 다음과 같은 공식에 의해 카이스퀘어 검정을 할 수 있다. 여기서 b와 c만 쓰였는데, 이는 heterozygote한 parents만을 고려하기 때문이다.
- TDT 테스트 결과 A와 a가 공평하게 transmission이 되지 않고 어느 한쪽에 치우쳐 있다면, 이 allele가 특정 disease와 관련된 snp일 수도 있다.
- TDT 테스트도 multiple hypothesis testing issue가 있다. 왜냐면 TDT는 모든 locus에 대해 allele의 우성/열성의 transmission이 공평한지 아닌지 테스트하기 때문이다. 따라서, 이를 고려해주어야 한다.
- 만약, 자녀가 여러명이고 질병이 있는 자녀와 없는 자녀로 나누어져 있을 때에는 자녀간 allele frequency를 비교하는 방법도 있다.
Case control studies
같은 나이, 성별, 인종 하에서 case인 집단의 SNP/haplotype marker과 control인 집단의 SNP/haplotype marker의 frequency를 비교하는 것이다. 아래의 표를 보면 각 genotype에 따른 (GG, GT TT) case 및 control의 count를 나타내는 표와, 이를 이용한 Observed allele counts와 expected allele counts 를 나타내는 표가 있다.
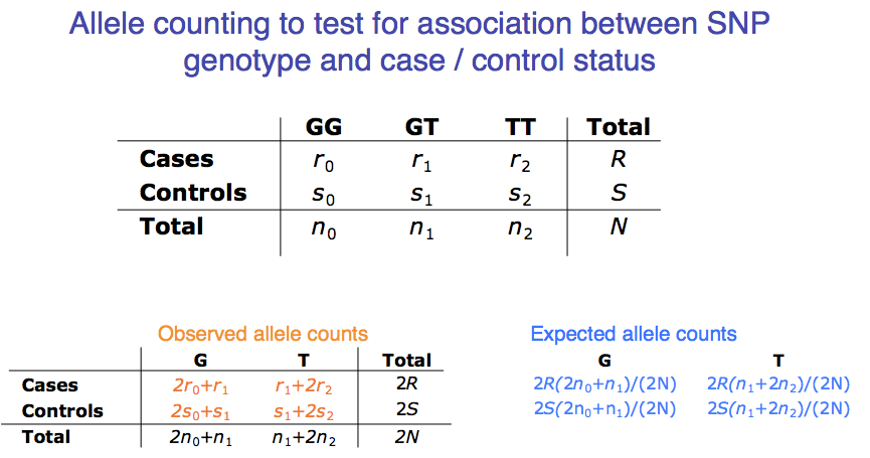
주어진 표를 이용하여 카이스퀘어 테스트를 수행할 수 있다. 카이스퀘어 검정의 특정 position의 allele가 case와 control간에 차이가 있는지를 테스트 한다. 다음의 예를 가지고 카이스퀘어 테스트를 수행해 보자.

우선 case 그룹의 allele A의 expected count 49가 어떻게 계산되었는지 알아보자.
마찬가지로 control 그룹의 allele a의 expected count 321은 다음과 같은 과정에 의해 계산되었다.
이제 카이스퀘어 검정을 위한 검정통계량을 계산해보자
에 의해 이다.
이 경우에도 multiple hypothesis testing을 고려해야 할까? 정답은 Yes이다. 왜냐하면 카이스퀘어 테스트가 모든 SNP에 대해 이루어지기 떄문이다.
Odds Ratio: Effect size를 측정하기 위한 도구
- Odds의 정의는 다음과 같다
- 예를 들어, 다음의 Allel count가 있는 경우를 생각해 보자.

- G allele에 해당하는 odds는 P(allele G at Cases) / P(allele G at Control) = a / c이다. 이 odds는 G allele가 case에 발생하는 odds이다.
- 마찬가지로 T allele에 해당하는 odds는 P(allele T at Cases) / P(allele T at Control) = b / d이다.
- Odds Ratio는 이 둘의 비를 뜻하므로 다음과 같다.
다음은 reference SNP ID number가 rs1333049인 genotype에서 심근경색과 association이 있는 allele들에대해 test를 수행한 결과이다.

- Odds ratio = 1이면, 질병과 이 allele (C, G)가 association이 없다,
- Odds ratio > 1이면 allele C가 질병의 위험성을 증가시킨다,
- Odds ratio < 1이면 allele C가 질병의 위험성을 감소시킨다 라고 해석할 수 있다.
- 여기서는 odds ratio = (2132 * 3089) / (1716 * 2783) = 1.38 > 1이므로 allele C가 심근경색의 위험을 증시킨다고 볼 수 있다. 오즈비에 대한 해석은 allele C를 가진 환자가 allele G를 가진 환자보다 심근경색에 대해 38% 더 큰 위험성을 가진다고 볼 수 있다.
- 또한 카이스퀘어 검정 결과의 p-value가 1.2*10E-13으로 매우 작으므로, allele C, G는 심근경색과 관련성이 매우 크다고 볼 수 있다.
- 여기서 multiple hypothesis testing도 고려해줘야 하는데, 왜냐하면 모든 position에 대한 allele를 카이스퀘어 검정하기 때문이다.
Reproducibility of Association Studies
대부분의 논문에서 보고된 association은 재분석 (reproduced) 되었을 때 일관성이 떨어진다. 이에 관련해 Hirschhorn et al (Genetics in Medicine, 2002)에서 association study를 review한 논문이 있는데, 이 논문에 따르면 603개의 association stduy 연구 결과 중 매우 적은 6개의 연구만이 reproduced되었다.
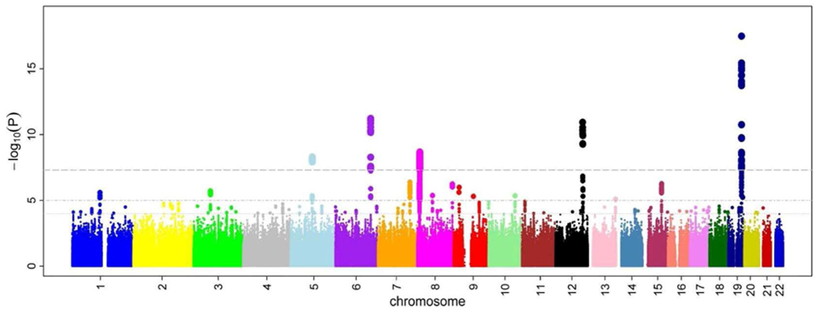
Size matters
다음의 왼쪽 그림은 각 SNP의 allele 대해서 카이스퀘어 검정을 통해 observed p-value와 expected p-value의 -log(p)값을 취한 후 그래프를 그린 것이다 (QQ plot). -log(expected p)보다 -log(observed p)가 큰 경우 그 SNP가 유효하다.
- 이러한 경우 p-value들 (multiple hypothesis와 관련된 어떤 종류의 p-value들)의 히스토그램을 그리면 uniform distribution이 되는데, 어떠한 SNP도 그래프에서 유효하지 않기 때문이다.
- 반면, p-value들의 히스토그램을 그렸을 때 uniform이 아닌 경우 significant한 SNP가 있다고 추측할 수 있다.
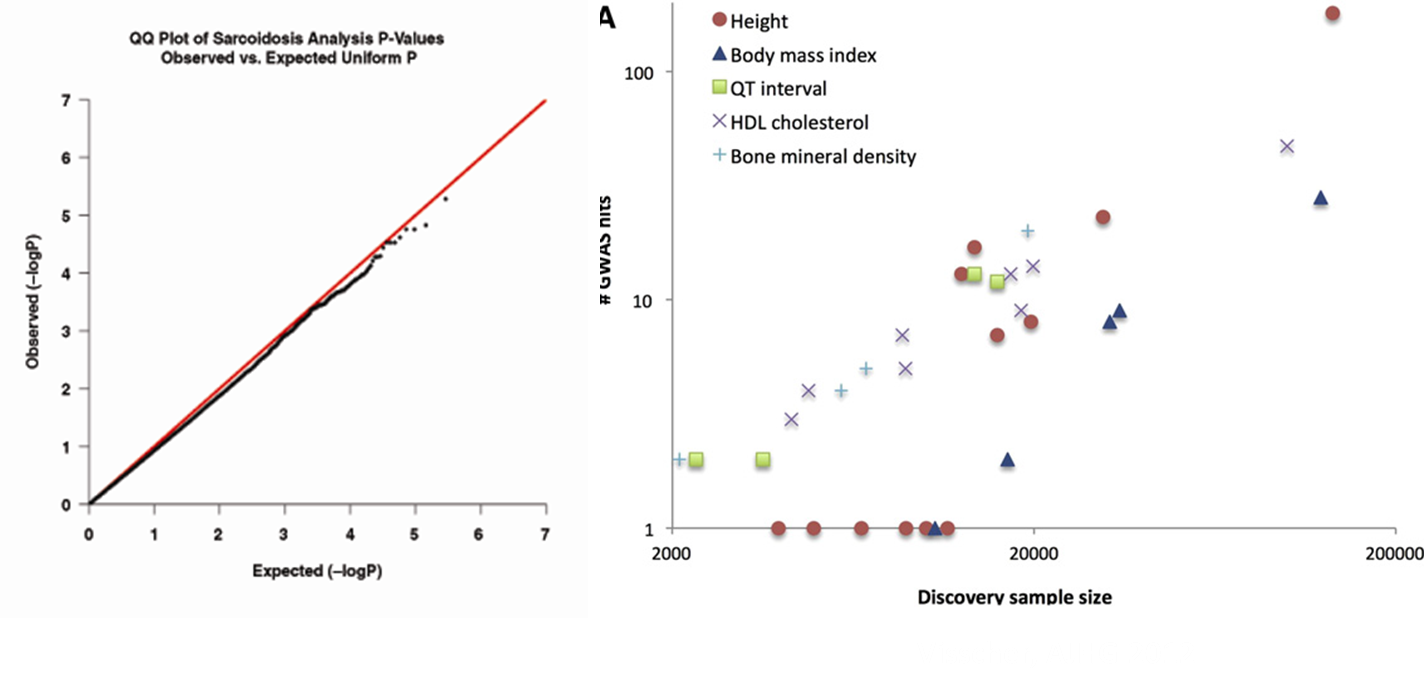
왼쪽 그림은 샘플 사이즈 (x축)별 GWAS에서 Hit한 SNP의 수를 (y축) study별로 그린 것이다. 그래프에서 볼 수 있듯이, 샘플 사이즈가 커질수록 Hit한 SNP의 수가 많아지는 것을 알 수 있다. 이는 샘플 사이즈가 커질 수록 effect size와 p-value를 더 정확하게 estimate할 수 있다는 의미이다.
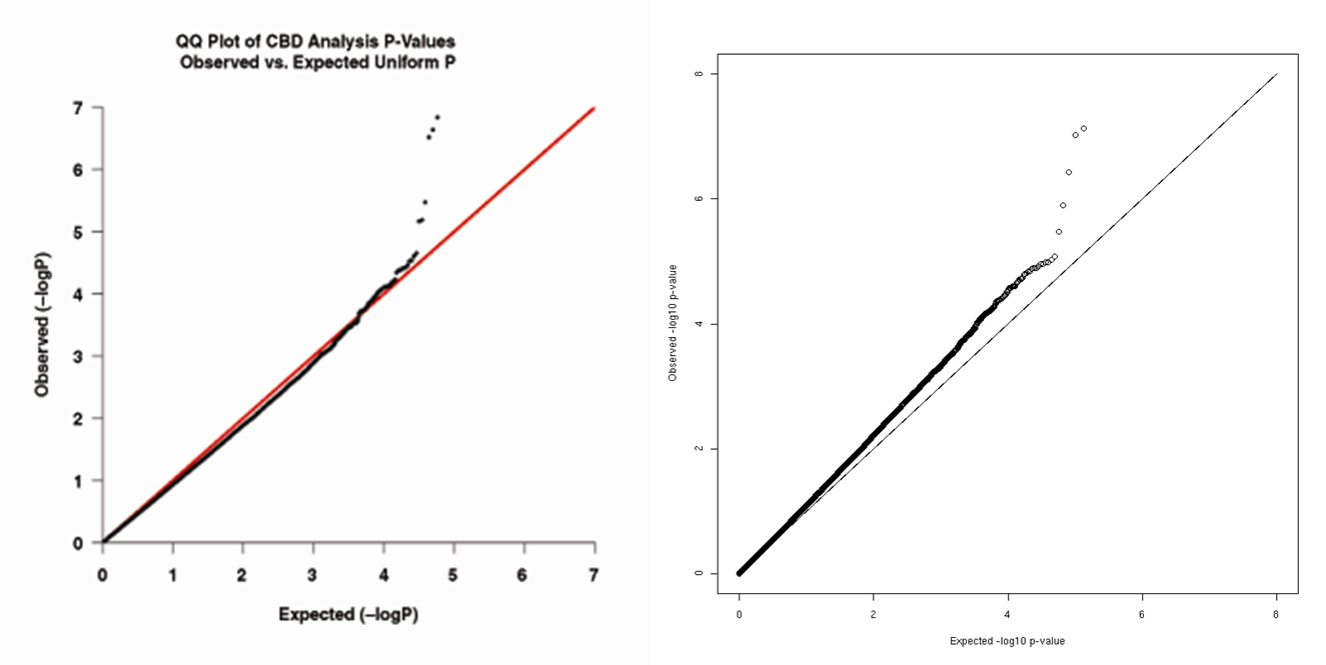
QQ Plot
다음의 GWAS study에 대한 QQ plot을 보면 왼쪽의 경우 좋은 GWAS study라고 할 수 있고, 오른쪽의 경우 population stratification이 존재하는 경우라고 볼 수 있다. (case와 control 간에 인종이 다른 경우)
Population stratification
Case와 control간에 population의 분포가 동일하지 않은 경우 정확한 비교가 어려울 수 있다. 예를 들면 어떤 SNP는 특정 인종에서만 나타나는 경우가 있다. 이러한 경우에는 case와 control간의 population분포가 동일하지 않으면 잘못된 결과를 도출하게 된다. 또한, population마다 disease frequency나 allele frequency가 다르기 때문에 population stratification을 확인하는 것은 중요하다.
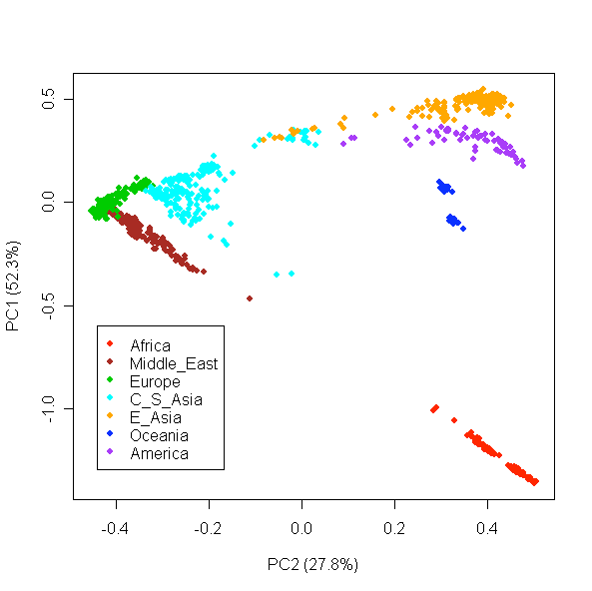
이러한 population stratification을 찾아내는 방법중의 하나가 모든 SNP에 대해 Principle Component Analysis (PCA)를 하는 것이다. 위의 그림은 특정 SNP에 대해 PCA를 한 후 첫번째 PC (PC1, x축)과 두 번째 PC (PC2, y축)을 그래프로 그린 것이다. 그림에서 여러 클러스터를 관측할 수 있는데 각 클러스터가 특정 인종을 나타낸다는 것을 알 수 있다.
분석을 진행할 떄에는 하나의 인종만을 선택하거나, 적어도 case와 control간의 population이 유사하도록 해야한다.




